Elektromagnetyczna transformacja DNA
Jeden z najkonkretniejszych jak dotąd eksperymentów demonstrujących główną rolę zakodowanego światłem włókna DNA przeprowadził Luc Montagnier, który w roku 2008 otrzymał Nagrodę Nobla w dziedzinie fizjologii i medycyny za odkrycie wirusa zespołu nabytego braku odporności (AIDS), zwanego wirusem HIV. W tym eksperymencie wykazano, że w kulturze, w której hodowano szczep patogenicznych bakterii, pozostał po ich całkowitym usunięciu emitowany przez nie szczególny sygnał elektromagnetyczny. Kiedy w środowisku tej kultury emitującym tę elektromagnetyczną częstotliwość umieszczono taki sam, ale nie patogeniczny, szczep, przekształcił się on w analogiczny patogeniczny szczep i zaczął emitować taki sam sygnał, co poprzedni. W ten sposób elektromagnetyczna częstotliwość przekształciła szczep, co oznacza, że oddziaływała na molekułę DNA nie patogenicznego szczepu i „przekodowała” ją.
Wszystkie biomolekuły tworzą kompleksy z wodą, która odgrywa zasadniczą rolę w funkcjonowaniu żywych molekuł. Woda nie jest bezczynnym ośrodkiem, w którym zachodzą reakcje biochemiczne, ale istotnym dla życia czynnikiem. Montagnier wysunął hipotezę mówiącą, że w wodnych nanostrukturach przechowywane są elektromagnetyczne częstotliwości – kompleksowe konformacyjne kompozycje makromolekularnej wody. Woda nie tylko przyjmuje unikalny wzorzec konformacji biomolekuł (i bezpośrednio wpływa na ich trójwymiarowy kształt w czasie ich formowania się), ale odpowiada również za większość ich elektrycznej aktywności za sprawą ich interakcji z jej momentem dipolowym.
Morficzny rezonans i konformacje DNA
Porównanie kodujących proteinę segmentów DNA – genów – różnych gatunków pokazuje istnienie wysokiego poziomu oszczędności oznaczającego, że całe życie korzysta z takiego samego – z niewielkimi różnicami – molekularnego kompletu narzędzi, czyli że geny są homologiczne. Te same geny, które rządzą molekularną i strukturalną maszynerią muszki owocówki, znajdujemy u człowieka. Te geny odpowiadają głównie za wytwarzanie molekularnej maszynerii, zaś wysoki stopień homologii i oszczędności dowodzi, że to nie one są źródłem różnic pomiędzy gatunkami i poszczególnymi osobnikami. Sekcje DNA, które odpowiadają za tworzenie tych różnic, są znane od dawna, ponieważ są wykorzystywane do identyfikacji gatunków i pojedynczych osobników. Polimorfizm długości fragmentów restrykcyjnych polega na badaniu pociętych przez enzymy restrykcyjne unikalnych odcinków DNA (polimorficznych segmentów), które określa się jako „odcisk palca DNA” i wykorzystuje do identyfikacji gatunków, podgatunków i kladów5 aż po pojedyncze osobniki. To powinno stanowić istotną wskazówkę. Jeśli poszukuje się tego, co sprawia, że jeden gatunek różni się od pozostałych, sekcje DNA używane do identyfikacji określonych gatunków są dobrym punktem wyjścia dla takich poszukiwań.
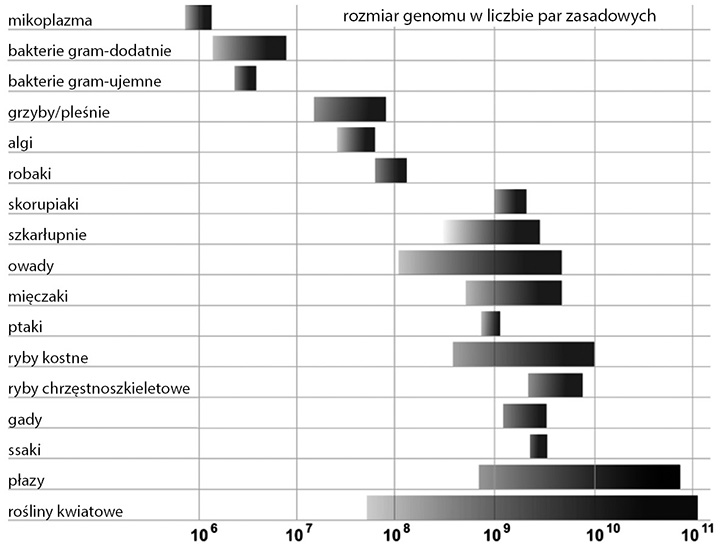
Te unikalne sekcje DNA są niekodującymi segmentami. Nazywanie tych sekcji genomu „śmieciowym DNA” to jak nazwanie silnika samochodu „zapasową oponą”. Jak ważne są niekodujące sekcje DNA? Jedną ze wskazówek powinna być ich dominacja w genomie. U ludzi od 95 do 98 procent genomu jest niekodująca! Podobnie jest u szeregu innych gatunków, przy czym między wzrastającą złożonością organizmu i ilością niekodującego DNA występuje korelacja. Na przykład bakterie, takie jak Escherichia coli (pałeczka okrężnicy), mają bardzo mało intergenicznego DNA. Ich genomy są architektonicznie uproszczone i stanowią proste okrągłe włókna, które nie tworzą złożonych konfiguracji, mimo iż liczą w przybliżeniu 4290 genów. Co ciekawe, niewielki nicień, Caenorhabditis elegans, posiada więcej genów niż ludzie. Jednak ludzie mają więcej niekodującego DNA, co pokazuje zależność między wielkością genomu i niekodującym DNA (spora część dużych rozmiarami genomów roślin wynika z poliploidów – wielokrotnych zestawów tych samych chromosomów wystarczających do wytworzenia różnorodności gatunkowej (specjacji) bez jakiejkolwiek zmiany w kodującym proteiny genomie). Trzy główne funkcje niekodujących segmentów, jakie dotychczas udało się zidentyfikować, to:
• Około połowa niekodujących regionów genomu składa się z ruchomych elementów genetycznych, które modulują ekspresję genów i mogą przemodelować chromosomy.
• Druga połowa składa się ze zmiennej liczby występujących w tandemie powtarzających się sekwencji, nazywanych satelitarnym DNA. Za sprawą określonych konformacyjnych aranżacji służą one jako interfejs łączący DNA z polem morficznym.
• Te dwie części intergenicznego DNA przejawiają ekspansję w genomie, która ma na celu zwiększanie pojemności biomolekuły do przenoszenia informacji.
Przemodelowywanie chromosomu
Mobilne elementy genetyczne stanowią wysoce funkcjonalną porcję niekodującego DNA. Umożliwiają DNA reagowanie na warunki środowiska i przemodelowywanie genomu poprzez aktywowanie transponowalnych elementów, które mogą translokować segmenty genomu i przemodelowywać chromosomy. Ponieważ regiony genomu są modularne, możliwa jest ich translokacja przy pełnym zachowaniu funkcjonalności, przy czym po takiej translokacji będą one inaczej wyrażane. Może to powodować przerywaną i szybką specjację – rodzaj praktycznie natychmiastowej ewolucji, ponieważ może do niej dojść w czasie życia jednego organizmu. Te segmenty DNA są aktywowane przez promieniowanie wysokiej częstotliwości i dlatego najczęściej do tych zmian dochodzi w miejscach jego występowania.
Porównanie genomów gatunków sklasyfikowanych w linii wielkich małp, takich jak ludzie i szympansy, pokazuje, że regiony genów kodujące proteiny są prawie identyczne, stąd różnice międzygatunkowe wydają się tkwić w niekodującym DNA. W szczególności retrotranspozony, zwane sekwencjami Alu, wykazują zdolność odgrywania wyjątkowej roli w ludzkim genomie, jako że są najliczniejszymi elementami, które występują w ponad milionie kopii.
Rozmiar genomu różnych organizmów wyrażony w liczbie nukleotydowych par zasadowych.
Tak więc modularna architektura DNA dostarcza logicznego wyjaśnienia, w jaki sposób transponowalne elementy mogą współuczestniczyć w przemodelowywaniu chromosomów. Do czego jednak służy satelitarne DNA, które zawiera proste sekwencje mogące powtarzać się setki razy? Wyjaśnienie pojawia się, kiedy rozpatruje się szczególną charakterystykę powtarzalnych sekwencji w świetle teorii rezonansu morficznego. Powtarzalne sekwencje mają zdolność tworzenia specjalnych powiązań, włącznie z trójskładnikowym i czteroskładnikowym pasmowym DNA, a także z szeregiem innych specyficznych elementów strukturalnych. Na przykład regiony telomeru6 są złożone z 3000 do 20 000 powtórzeń par zasadowych sekwencji TTAGGG. Ten bogaty w G region tworzy to, co jest nazywane G-quadruplex (G4-DNA) i może tworzyć czterowłóknowe kompleksy DNA.
Te różne konformacje tworzone przez satelitarne DNA wchodzą w specyficzne rezonanse i polem morficznym i są zdolne dostroić się do bardzo specyficznych programów informacyjnych. Satelitarne DNA są wysoce specyficzne dla każdej jednostki, co oznacza, że każda jednostka dostraja się do wyraźnego morfogenetycznego modelu, który jest dla niej unikalny. Ponadto ze względu na dużą plastyczność tego polimeru – zdolnego do gwałtownego przejścia przez wiele konformacji w molekule DNA – może on służyć do modulowania zachowań komórki, tkanki i organizmu poprzez zmianę rezonansu wzorców morficznych. Co więcej, zdolność satelitarnego DNA do zwiększania liczby powtarzalnych sekwencji zwiększa pojemność molekuły DNA, która pozwala na przenoszenie większej ilości informacji.